El Array CytoScan 750K permite detectar alteraciones en el número de copias para la mayoría de las regiones, descritas y no descritas, indicados especialmente en el área de Pediatría, Neuropediatría y Ginecología, obteniendo de esta forma más información genómica en una sola prueba.
Tanto es así que es recomendada como la primera estrategia diagnóstica en la discapacidad intelectual y en la población pediátrica con rasgos dismórficos o anomalías múltiples congénitas, permitiendo la identificación de nuevos síndromes, un mejor conocimiento de los mismos y una mejor asistencia sanitaria del paciente.
A efectos de detección de dichas alteraciones, el uso de arrays incrementa, al menos en un 10%, la capacidad de detección de alteraciones en los casos con dismorfias o discapacidad intelectual y cariotipo normal, constituyendo una alternativa de diagnóstico más eficiente.
El trastorno del espectro autista (TEA) es un conjunto de trastornos del desarrollo neurológico clínicamente heterogéneo caracterizado por un fenotipo conductual complejo que incluye déficits en las funciones sociales y/o cognitivas y que afecta al 1% de la población infantil con una mayor frecuencia en hombres (4:1).
La causa del TEA no se conoce con exactitud, siendo la alteración multigénica la causa más probable. A pesar de ello, se estima que hasta un 20-30% de los individuos puedan tener una etiología genética específica y diagnosticable. En este sentido, los avances en la tecnología de microarrays y secuenciación masiva han hallado asociación entre TEA y diferentes alteraciones de número de copia (CNV) y polimorfismos de un solo nucleótido (SNP) de genes con un papel importante en la formación de sinapsis y la función.
Por ello, gracias a las nuevas herramientas de diagnóstico y a un mayor conocimiento de la enfermedad, en los últimos años ha habido un aumento en la prevalencia del TEA.
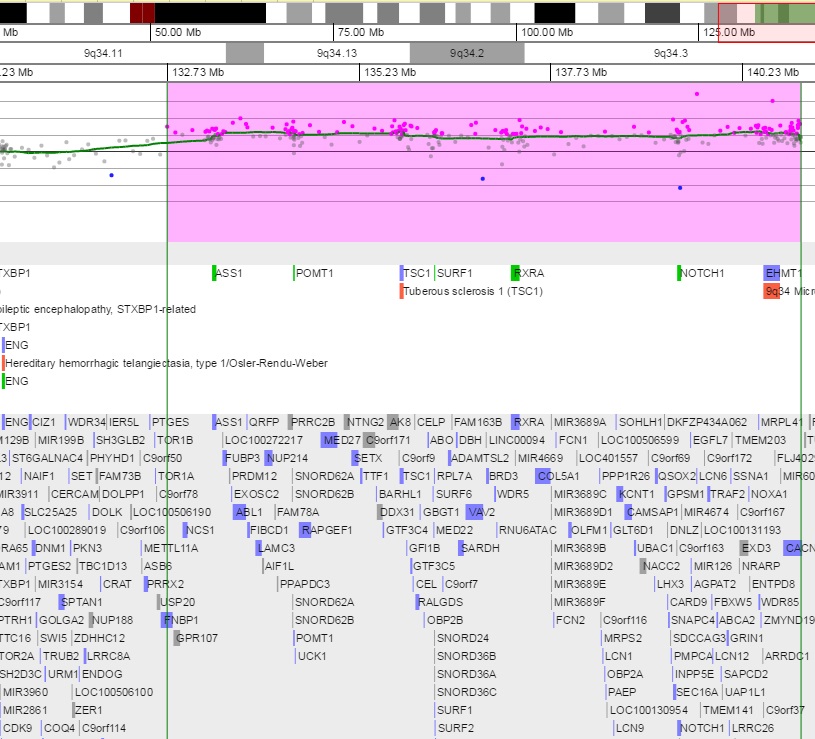
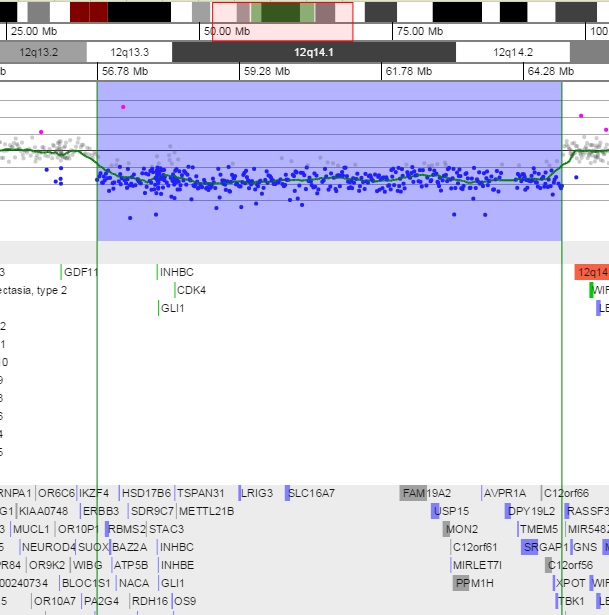
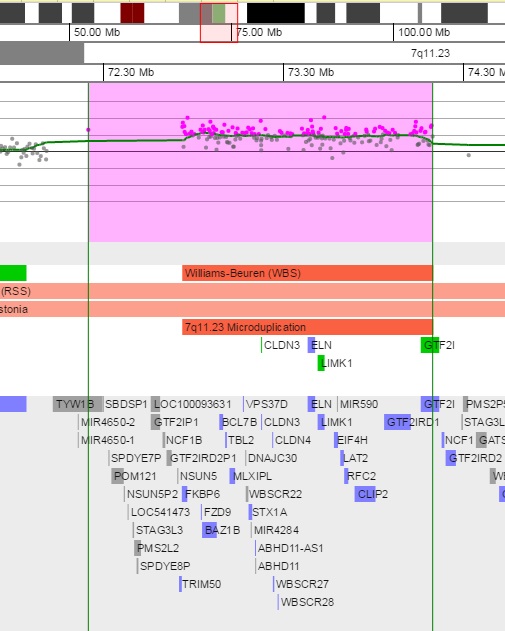
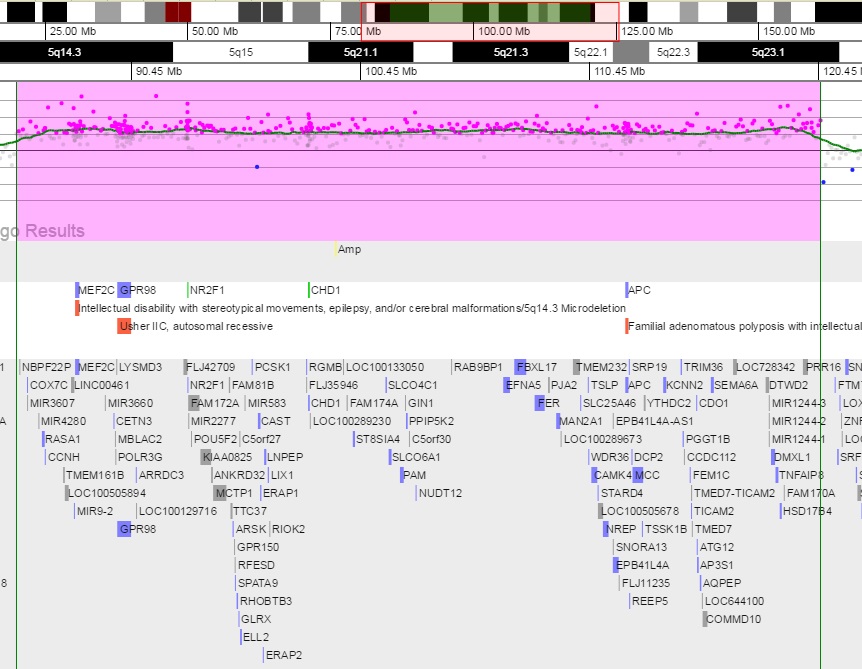
EJEMPLOS DE CASOS ANALIZADOS:
SIGNIFICACIÓN CLÍNICA DE LAS CNV*:
-CNV patogénica. Variante con asociación clínica a un fenotipo claro e identificado.
-CNV benigna. Variante presente en población sana y descrita como tal en bases de datos.
-CNV de significado incierto. CNV que no aparece identificada claramente como benigna o patogénica en el momento del análisis. Puede ser: a) probablemente patogénica, si implica genes cuya función podría explicar un evento patológico; b) probablemente benigna, si está heredada de un progenitor sano y no existe referencia patológica o afecta parcialmente a una región típicamente polimórfica; c) de significado incierto, si no existe evidencia médica que indique una de las anteriores.
*Según las propuestas señaladas en el Consenso para implementación de los Arrays (CGH y SNP-arrays) en la Genética Clínica, Instituto Roche 2012 (www.institutoroche.es ) y de Vermeesch J et al. Hum Mutat 2013;33(6):906-915.